細胞の中にはおよそ2万個の遺伝子が含まれますが、これらは単独で働くのではなく、お互いに制御しあうことで活性と不活性(遺伝子発現)を切り替えています。遺伝子間の制御関係は、複雑なネットワークを形成することが分かってきています。このネットワークに基づいて細胞の性質が決まったり、あるいは性質の切り替えが起きたりすることから、遺伝子制御ネットワークは「細胞の設計図」と言えます。しかし、遺伝子制御ネットワークは未だ部分的にしか明らかになっておらず、また正確にそれを決定することは困難な課題でした。
石川雅人 医生物学研究所特定助教、望月敦史 同教授、遊佐宏介 同教授、永樂元次 同教授と、木立尚孝 東京大学准教授らの共同研究グループは、様々な遺伝子をノックアウトした後の時系列発現変化を計測したデータから、数理モデルに基づいて遺伝子制御ネットワークを高精度かつ半自動的に推定する手法RENGEを開発しました。シミュレーションデータとヒトiPS細胞における発現計測データの両方において、RENGEは既存手法を凌駕する性能を示しました。RENGEにより推定されたヒトiPS細胞の遺伝子制御ネットワークの解析から、iPS細胞の多能性を維持する上で重要なタンパク質複合体が示唆されました。RENGEにより推定された遺伝子制御ネットワークを利用することで、細胞分化の鍵因子をネットワークから決定し、細胞運命制御を実現できる可能性があります。
本研究成果は、2023年12月28日に、国際学術誌「Communications Biology」にオンライン掲載されました。
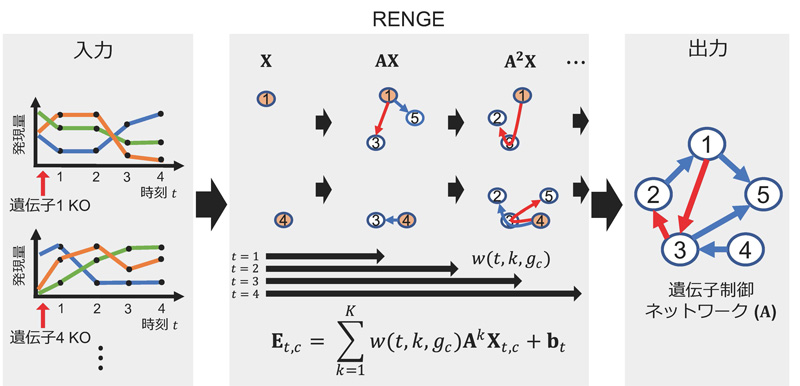
「新しい実験技術の開発により取得可能となった大規模データから、どのように役に立つ情報を抽出するかは現在の生命科学の大きな課題です。本研究では、遺伝子ノックアウト後の発現ダイナミクスを説明する数理モデルを用いることで、遺伝子制御ネットワークを他手法より正確に推定できました。新しい実験技術とそれに適した数理的解析手法を組み合わせることで、生命システムを効率的に解明していくことができると考えられます。」(石川雅人)
【DOI】
https://doi.org/10.1038/s42003-023-05594-4
【KURENAIアクセスURL】
http://hdl.handle.net/2433/286528
【書誌情報】
Masato Ishikawa, Seiichi Sugino, Yoshie Masuda, Yusuke Tarumoto, Yusuke Seto, Nobuko Taniyama, Fumi Wagai, Yuhei Yamauchi, Yasuhiro Kojima, Hisanori Kiryu, Kosuke Yusa, Mototsugu Eiraku & Atsushi Mochizuki (2023). RENGE infers gene regulatory networks using time-series single-cell RNA-seq data with CRISPR perturbations. Communications Biology, 6:1290.





