ターゲット
公開日
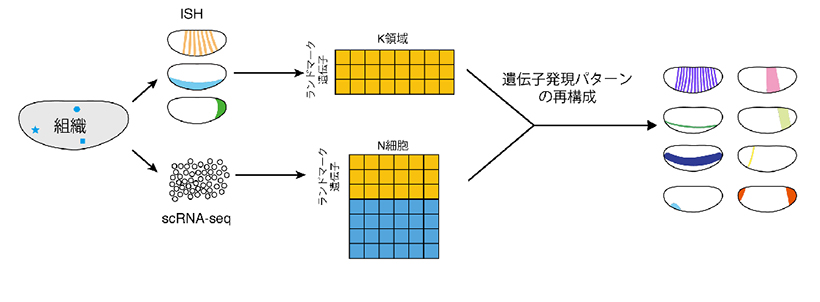
本田直樹 生命科学研究科特命教授(兼・広島大学教授、生命創成探究センター客員教授)、近藤武史 同特定助教、大河内康之 医学部生、坂口峻太 生命科学研究科・日本学術振興会特別研究員、中江健 情報学研究科特定助教らの研究グループは、1細胞RNAシーケンシング(RNA-seq)法で計測された遺伝子発現データから、遺伝子発現の空間的パターンをあたかもパズルを解くかのように再構成する機械学習法(Perler)を開発しました。
この提案手法は、従来法と比べて高精度かつ頑健に空間的遺伝子発現パターンを再構成することが示されました。正確な空間遺伝子発現パターンの再構成が実現されたことにより、発生過程における形作りや多細胞からなる組織機能の理解にも貢献することが期待されます。さらに、再生医療として作成されたミニ臓器が、実際の体内臓器をどの程度再現しているのかなどの評価に応用されることが期待されます。
本研究成果は、2021年6月17日に、国際学術誌「Nature Communications」に掲載されました。
研究者情報
書誌情報
【DOI】https://doi.org/10.1038/s41467-021-24014-x
【KURENAIアクセスURL】http://hdl.handle.net/2433/263916
Yasushi Okochi, Shunta Sakaguchi, Ken Nakae, Takefumi Kondo, Honda Naoki (2021). Model-based prediction of spatial gene expression via generative linear mapping. Nature Communications, 12, 3731.





