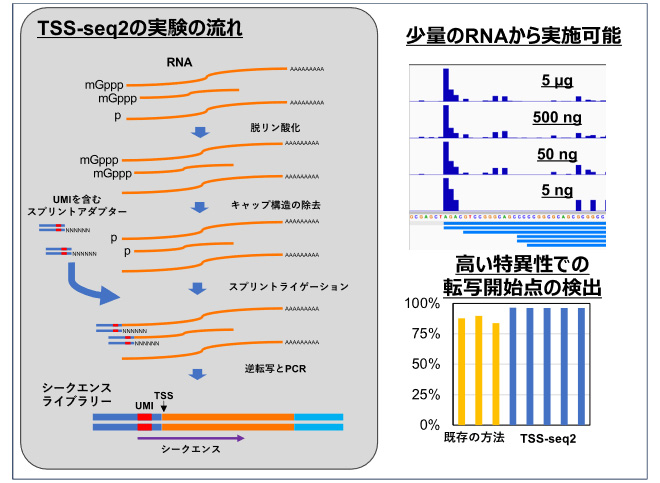
松下智直 理学研究科教授、野田口理孝 同教授(兼:名古屋大学特任教授)、関真秀 東京大学特任准教授、鈴木穣 同教授、吉田聡子 奈良先端科学技術大学院大学教授、壽崎拓哉 筑波大学准教授、黒谷賢一 名古屋大学特任准教授らの研究グループは、ゲノムDNAから遺伝子を読み取る開始位置である転写開始点(TSS)を網羅的に決定するTSS-seq2法を開発しました。さらに、コシオガマ、ベンサミアナタバコ、ミヤコグサ、ハクサンハタザオの4種類の植物について、TSS情報の収集を行いました。
TSSの決定は、RNAの正確な構造や、TSSの近辺に存在して遺伝子の機能を調節する重要な領域であるプロモーター領域を同定するために重要です。正確性の高い転写開始点検出法は、必要なRNAの量が多く、プロトコルが複雑であるなど、簡単には実施できない手法が主流でした。今回、先行研究により開発されたTSS-seqを改良・簡略化することで、TSS-seq2を開発しました。TSS-seq2は既存の方法よりも特異的に転写開始点を検出でき、5ナノグラムと少量のRNAからでも実施できます。
今回開発した方法は、様々な生物種や組織でのmRNAの正確な構造の同定や遺伝子の制御の研究、特に、希少な細胞種や微小な組織などの少量のサンプルの解析への応用が期待されます。また、今回収集した植物のTSS情報は、これらの植物種の研究の基盤データとなることが期待されます。
本研究成果は、2023年11月22日に、国際学術誌「Nucleic Acids Research」にオンライン掲載されました。
【DOI】
https://doi.org/10.1093/nar/gkad1116
【KURENAIアクセスURL】
http://hdl.handle.net/2433/286793
【書誌情報】
Masahide Seki, Yuta Kuze, Xiang Zhang, Ken-ichi Kurotani, Michitaka Notaguchi, Haruki Nishio, Hiroshi Kudoh, Takuya Suzaki, Satoko Yoshida, Sumio Sugano, Tomonao Matsushita, Yutaka Suzuki (2024). An improved method for the highly specific detection of transcription start sites. Nucleic Acids Research, 52(2):e7.