野外の生物集団において、在来生物種と交雑の恐れがある外来種や、害虫の殺虫剤抵抗性系統がどの程度の比率で存在するかを把握するために、各個体からDNAを抽出してPCR検査等で遺伝子型を決定する、個体別遺伝子診断が従来行われてきました。しかし、比率を知りたい対立遺伝子(アリル)をもつ個体が集団中に稀にしか存在しない場合は、推定精度を保つために数十~数百個体以上を診断する必要がありました。そこで実験操作の回数を減らすため、数個体ずつまとめてDNA抽出された「バルクサンプル(混合DNA溶液)」による診断で、対立遺伝子頻度を推定する方法が模索されてきました。
バルクサンプルのDNA含有量や、対立遺伝子ごとの含有比は、定量PCRや量的DNAシーケンシング等の手法によって測定できます。もし各個体が同じ量のDNAを持つならば、バルクサンプル中のDNAの比率は、そのままバルクサンプルに含まれる個体の遺伝子構成を示します。しかし現実には、各個体の体を構成する細胞の数によってDNA量が異なることに加え、DNA量は死後の分解によっても減少します。そのためトラップで捕獲した個体からバルクサンプルを作ってDNA量の比を測定すると、野外の個体の存在比から大きくずれる場合があります。
刑部正博 農学研究科准教授(現:名誉教授)は、農研機構、宇都宮大学らと共同で、各個体から得られるDNA量のばらつきを「ガンマ分布」という確率分布で近似することにより、生物集団における対立遺伝子の比率を、その推定値がどの程度確からしいかの指標(信頼区間)とともに推定できる統計モデルを開発しました。複数のバルクサンプルが用意され、その各々が何個体から構成されるかが分かっていれば、適用が可能です。
本モデルを定量PCR解析に適用して、対立遺伝子の比率とその信頼区間を簡便に求められるようにするべく、フリーの統計解析環境であるRのためのパッケージ“freqpcr”を開発し公式サイトで配布しています(https://cran.r-project.org/package=freqpcr)。すでに本パッケージは、ミカンハダニにおける殺ダニ剤抵抗性遺伝子の地域分布パターンの解析をはじめ、野外で稀な遺伝子の存在比率をより少ない検査回数で高精度に推定する目的で活用されています。また農業害虫のみならず、希少生物種の保全や外来種・系統の侵入警戒を目的としたモニタリングなどにも役立ちます。
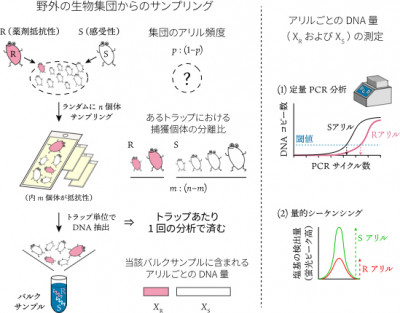
図:野外の生物集団からのサンプリング
(左)ある単数体生物の集団が、薬剤抵抗性のアリル(R)を頻度pで持っており、残りの1−pが感受性アリル(S)だったとします。粘着板トラップでn個体を採集したとき、抵抗性個体がm個体含まれる確率は二項分布で表せます。このトラップから一括抽出されたDNA溶液(バルクサンプル)に含まれる、アリルごとのDNA量(XR, XS)はm個体ないしn−m個体分の合計量です。
(右)アリルごとのDNA量を測定して比率を求めるために、定量PCR分析の増幅サイクル数(Cq値)や、量的DNAシーケンシングの塩基検出量といった指標が用いられます。トラップあたり1回の分析でよいため労力を削減できますが、DNA量の個体差があるため含有比の測定値がm:(n−m)とは厳密には一致せず、統計理論に基づいた補正が必要となります。





