三木邦夫 名誉教授、竹田一旗 理学研究科准教授、玉田太郎 量子科学技術研究開発機構チームリーダー、花園祐矢 同博士研究員(現:東京医科歯科大学助教)、平野優 同主幹研究員、日下勝弘 茨城大学教授は共同で、光合成細菌の光合成電子伝達を担うタンパク質である高電位鉄イオウタンパク質(HiPIP)の高精度全原子結晶構造解析に成功しました。水素原子の観察に優れた中性子の特徴を生かして水素原子の位置を精密に決定し、タンパク質の立体構造形成に大きく影響するペプチド結合の平面構造について新たな構造モデルを提唱しました。
生命活動の中心的役割を担うタンパク質は、数十から数万のアミノ酸がペプチド結合により鎖状につながった分子です。一般にペプチド結合は平面構造が最も安定であり、タンパク質分子中のペプチド結合もすべて同じ平面構造であるとの仮定の下で、これまではタンパク質の構造が議論されてきましたが、その仮定を実際に確かめた研究例はありませんでした。なぜなら、タンパク質分子を構成する原子の約半分が水素原子のため、タンパク質の構造を決定する際に主に用いられるX線結晶構造解析という手法だけでは、高い精度で観測することができないためです。
そこで、研究グループは水素原子を直接観察できる中性子結晶構造解析という手法に着目しました。この解析を行うためには、極めて高品質かつ、通常X線結晶構造解析で用いられるサイズの約1万倍という大きな結晶試料を作成することが必要でしたが、これらの課題を克服し、水素原子を直接観測することができました。その結果、ペプチド結合はすべて同じ平面構造を取るという仮定に基づいた従来モデルを用いることなく、HiPIPの全原子構造を1.2Å(オングストローム。1Åは100億分の1メートル)分解能という極めて高い解像度で決定することに成功しました。その結果、ペプチド結合は周囲の環境によって多様な構造をしていることを世界で初めて明らかにし、ペプチド結合の新たなモデルを提唱しました。
今回明らかにしたペプチド結合の構造についての知見は、電子伝達機能などタンパク質の機能発現のメカニズムを理解する上で重要です。また、今回提唱する新しいモデルに基づいてタンパク質の立体構造を精密に捉えることで、生命科学研究の進展に貢献し、研究で得られた知見は、医学、薬学、工学分野に幅広く応用されることが期待できます。
本研究成果は、2022年5月21日に、科学誌「Science」の姉妹誌である「Science Advances」のオンライン版に掲載されました。
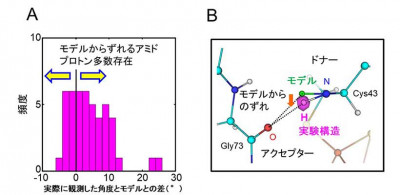
A:ドナー原子とアクセプター原子間の角度(∠N-H...O)の実験データとモデルとの差を示す。モデルからずれるアミドプロトンが多数存在し、モデルより大きい角度(右矢印)の頻度が高いことがわかる。
B:実測した水素原子(実際は重水素原子)。マゼンタが原子核散乱長密度分布で水素(重水素)原子を除いて計算したもの。モデル(緑)に比べて角度は大きく、アクセプターの方を向いている。
【DOI】
https://doi.org/10.1126/sciadv.abn2276
【KURENAIアクセスURL】
http://hdl.handle.net/2433/274106
【書誌情報】
Yuya Hanazono, Yu Hirano, Kazuki Takeda, Katsuhiro Kusaka, Taro Tamada, Kunio Miki (2022). Revisiting the concept of peptide bond planarity in an iron-sulfur protein by neutron structure analysis. Science Advances, 8(20):eabn2276.





