菅野茂夫 理学研究科研究員、松永幸大 東京理科大学教授、藤本聡 同研究員、桑田啓子 名古屋大学特任助教、刑部敬史 徳島大学教授の研究グループは、ゲノム編集技術を利用して、植物のDNAを生きたまま解析するTALE-FP法の開発に成功しました。本研究成果は、ゲノム育種により作成された品種選抜の高速化、植物における染色体異常の検出の簡便化など、農作物研究に大きく貢献することが期待されます。
本研究成果は2016年10月7日に英国の科学雑誌「Journal of Experimental Botany」電子版に掲載されました。
研究者からのコメント
本研究成果により、植物のDNAをあらゆる組織で生きたまま解析することができるようになりました。染色体標本を作製することなく、DNAの分布を解析できることから、ゲノム育種の研究現場における染色体解析や倍数性解析の効率化を図ることが可能になります。本法に用いたTranscription activator like effector(TALE:DNA配列に特異的に結合するDNA結合タンパク質)は無毒化されており、どの植物にも導入可能なタンパク質であるため、今後、有用作物のDNA解析にも応用可能であると考えられます。
概要
植物のDNAを解析するためには、植物細胞を破砕して、細胞核からDNAを抽出する必要がありました。また、植物の染色体の解析には蛍光in situハイブリダイゼーション法という手法が主に用いられていましたが、細胞を固定する必要があるため、生きた植物のDNAをリアルタイムで解析する手段はありませんでした。近年、DNAの細胞核の中での3次元的な位置が、DNA情報が読み取られる頻度に影響を及ぼすことが分かってきていますが、時々刻々と変わる位置情報を観察する手法が限られるため、新たな手法の開発が急務でした。
そこで本研究グループは、モデル植物であるシロイヌナズナを用いて、植物のDNAを生きた植物の組織や器官で検出するTALE-FP法を開発しました。ゲノム編集技術で用いられているキサントモナス菌由来のTALEのDNA結合ドメインに蛍光タンパク質を連結させたTALE-FPを植物体内で発現させました。
その結果、特定のDNA配列の動態を植物のさまざまな組織や器官で解析することに成功しました。これにより、特定の分裂組織から染色体標本を作製することなく、DNA配列の位置や分布を検出することが可能になりました。
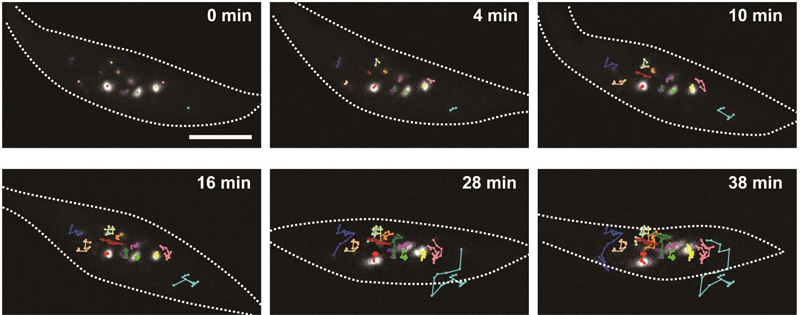
図:細胞内を動き回るTALE-FP
白い点線は細胞核を示す。細胞核内をテロメア(染色体末端部分の構造)を認識するTALE-FPが動き回っていることがわかる。TALE-FPのそれぞれの輝点をトラッキングしたラインを示している。
詳しい研究内容について
書誌情報
【DOI】
http://dx.doi.org/10.1093/jxb/erw371
【KURENAIアクセスURL】
http://hdl.handle.net/2433/217423
Satoru Fujimoto, Shigeo S. Sugano, Keiko Kuwata, Keishi Osakabe and Sachihiro Matsunaga. (2016). Visualization of specific repetitive genomic sequences with fluorescent TALEs in Arabidopsis thaliana. Journal of Experimental Botany. 67(21): 6101-6110.





