岡田大瑚 医学研究科 博士課程学生、山田亮 同教授は、複雑な確率分布に幾何的な座標を与える新規の統計手法であるDEEF法を開発し、細胞集団プロファイルの違いや変化を定量・可視化することに成功しました。
生体内の細胞1つ1つのタンパク質や遺伝子の発現量はどれも同じではなく少しずつ違っているため、その細胞集団全体としての性質はこれらの細胞集団が作る分布として特徴づけることができます。このような分布は、細胞1つ1つに発現している遺伝子やタンパク質量を多数の細胞について測定する1細胞発現解析によって測定することができます。一方で、情報科学の分野では、確率分布同士の違いを幾何学的に研究する情報幾何学が発展してきました。
本研究では、1細胞発現データのような複雑な分布に対して情報幾何的な座標を与え、ぞれぞれの座標が説明するパターンを抽出する新しい統計手法を開発し、細胞集団プロファイルの違いや変化を低次元空間上で定量・可視化することに成功しました。この手法の応用例としては、経時的に観測したフロサイトメトリーデータから、観測時刻と観察時刻の間の分布状態を推測し、動画を再構成することが挙げられます。
本研究成果は、2020年4月11日に、国際学術誌「PLOS ONE」のオンライン版に掲載されました。
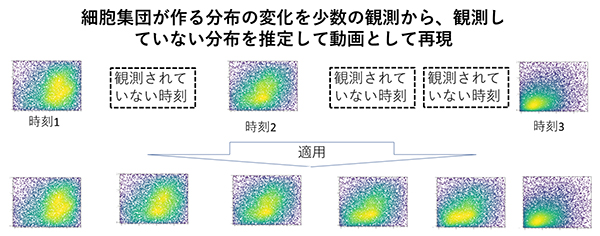
図:本研究の概要図
詳しい研究内容について
書誌情報
【DOI】 https://doi.org/10.1371/journal.pone.0231250
【KURENAIアクセスURL】 http://hdl.handle.net/2433/250313
Daigo Okada, Ryo Yamada (2020). Decomposition of a set of distributions in extended exponential family form for distinguishing multiple oligo-dimensional marker expression profiles of single-cell populations and visualizing their dynamics. PLOS ONE, 15(4):e0231250.





