鎌田真由美 医学研究科 准教授、荒木望嗣 同特定准教授、奥野恭史 同教授、安田浩之 慶應義塾大学 専任講師、浜本純子 同特任助教、池村辰之介 同助教、副島研造 同教授、土原一哉 国立がん研究センター先端医療開発センター 分野長、小林進 同分野長、 後藤功一 同 東病院科長、松本慎吾 同医長、 河野隆志 同 研究所分野長らのグループは、LC-SCRUM-Japanで構築した日本最大のがん臨床ゲノムデータを活用し、スーパーコンピュータ「京」を用いた予測システムにより、肺がんの遺伝子変異に対する薬剤有効性が高精度に予測可能なことを確認しました。
がんゲノム医療の普及により、さまざまな遺伝子の変異が同定され、治療薬(分子標的薬)の効果が予測されていますが、稀な遺伝子変異に対しては投薬効果の予測が難しく、薬剤を選ぶ上で大きな障害となっていました。
本研究グループでは、日本人の肺がんで最も多く変異の見られるEGFR遺伝子に注目し、約2,000例の肺がんの遺伝子変異を分析しました。その結果、稀なEGFR遺伝子変異をもつ肺がんに対して治療効果の高い抗がん剤をスーパーコンピュータ「京」を用いて高精度に予測することができました。
超高速・高性能な計算機を用いた本システムを実用化することで、より多くの肺がん患者に迅速に、有効性が高い治療薬を選ぶことが可能になると期待されています。また、他の多くの遺伝子にも適応を拡大することで、がんゲノム医療の進歩に大きく貢献することが予想されます。
本研究成果は、2019年5月1日に、国際学術誌「Proceedings of the National Academy of Sciences of the United States of America(PNAS)」のオンライン版に掲載されました。
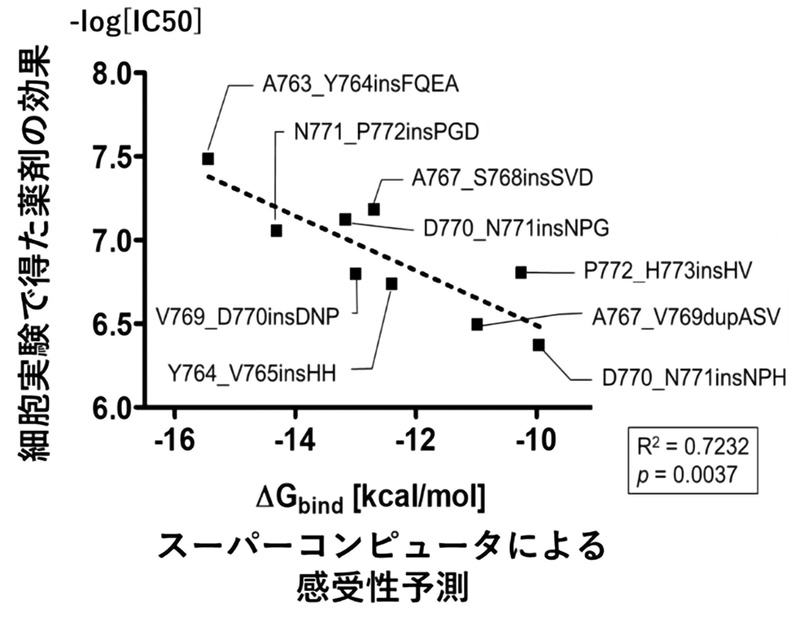
図:実験データと予測データの相関図。実験で得た薬剤の効果と予測データの間に高い相関が認められた。
詳しい研究内容について
書誌情報
【DOI】 https://doi.org/10.1073/pnas.1819430116
【KURENAIアクセスURL】 http://hdl.handle.net/2433/241348
Shinnosuke Ikemura, Hiroyuki Yasuda, Shingo Matsumoto, Mayumi Kamada, Junko Hamamoto, Keita Masuzawa, Keigo Kobayashi, Tadashi Manabe, Daisuke Arai, Ichiro Nakachi, Ichiro Kawada, Kota Ishioka, Morio Nakamura, Ho Namkoong, Katsuhiko Naoki, Fumie Ono, Mitsugu Araki, Ryo Kanada, Biao Ma, Yuichiro Hayashi, Sachiyo Mimaki, Kiyotaka Yoh, Susumu S. Kobayashi, Takashi Kohno, Yasushi Okuno, Koichi Goto, Katsuya Tsuchihara, and Kenzo Soejima (2019). Molecular dynamics simulation-guided drug sensitivity prediction for lung cancer with rare EGFR mutations. Proceedings of the National Academy of Sciences of the United States of America, 116(20), 10025-10030.





