藤本明洋 医学研究科特定准教授を中心とする研究グループは、がんの網羅的な遺伝子変異データを新たに開発した手法で解析し、がんの発生・進展に重要な役割を果たす遺伝子(以下、ドライバー遺伝子)候補である106の遺伝子を検出しました。このうちの約半数は既知のドライバー遺伝子であり、新たなドライバー遺伝子発見につながる可能性があります。
本研究成果は5月26日、Springer Nature社の学術誌 Scientific Reports に掲載されました。
研究者からのコメント
今回の研究で開発した手法は、従来の方法では見逃されていたドライバー遺伝子の発見や、がんの突然変異の生物学的意義の解明に貢献すると考えています。今後は、手法の改良とともに、発見されたドライバー遺伝子や突然変異の実験的検証を行いたいと考えています。
概要
近年のシークエンス技術の発展により、がんのゲノム配列決定が可能になりました。創薬研究の更なる発展を目的として、超並列シークエンサー(いわゆる次世代シークエンサー)を用いて、がんの突然変異の包括的カタログを作成するプロジェクト(ICGC; 国際がんゲノムコンシーアムやTCGA; The Cancer Genome Atlas計画などのプロジェクト)が進行しています。これらのプロジェクトによって、既に15,000症例を超えるがんの網羅的遺伝子変異データが公開されています。
これらのゲノム情報を解析し、ドライバー遺伝子を検出することが、がんゲノム研究の最も重要な問題の一つです。ドライバー遺伝子の特徴の一つは、変異がタンパク上の特定の部分に集積していることです。例えば、KRAS遺伝子ではコドン12、EGFR遺伝子ではコドン858や719に変異が集積することが知られています。
したがって、変異の集積を検出することで、ドライバー遺伝子の検出が可能であると考えられます。このような発想に基づき、DNA配列上(タンパクの1次構造上の)の変異の集積は、よく検討されています。
今回の研究では、タンパク質の立体構造を考慮することにより、DNA配列の解析では検出できない変異の集積を検出できるのではないかと考え、立体構造を考慮して変異の位置の偏りをテストする方法(3Dpermutation法)を開発しました(図)。
この方法では、立体構造が決定されている遺伝子について、立体構造内の変異間の距離を求め、並べ替え法(permutation法)で統計的有意性を検定します。この方法をTCGA計画で公開された21種のがんのデータに適用したところ、106遺伝子に有意な変異の偏りが検出され、ドライバー遺伝子候補と考えられました。これらのうち、約半数の遺伝子が既知のドライバー遺伝子( TP53, PIK3CA, PTEN, CDKN2A など)であり、本方法はドライバー遺伝子の検出に成功していると考えられました。
また、既知のがん関連遺伝子に加えて、クロマチン制御遺伝子( MLL3 )、DNA修復遺伝子( PAR G)、RNAスプライシン関連遺伝子( DHX9、SF1 )、ユビキチン化関連遺伝子( CUL1 )、転写因子( KLF6、EEF1B2 )などが検出されました。
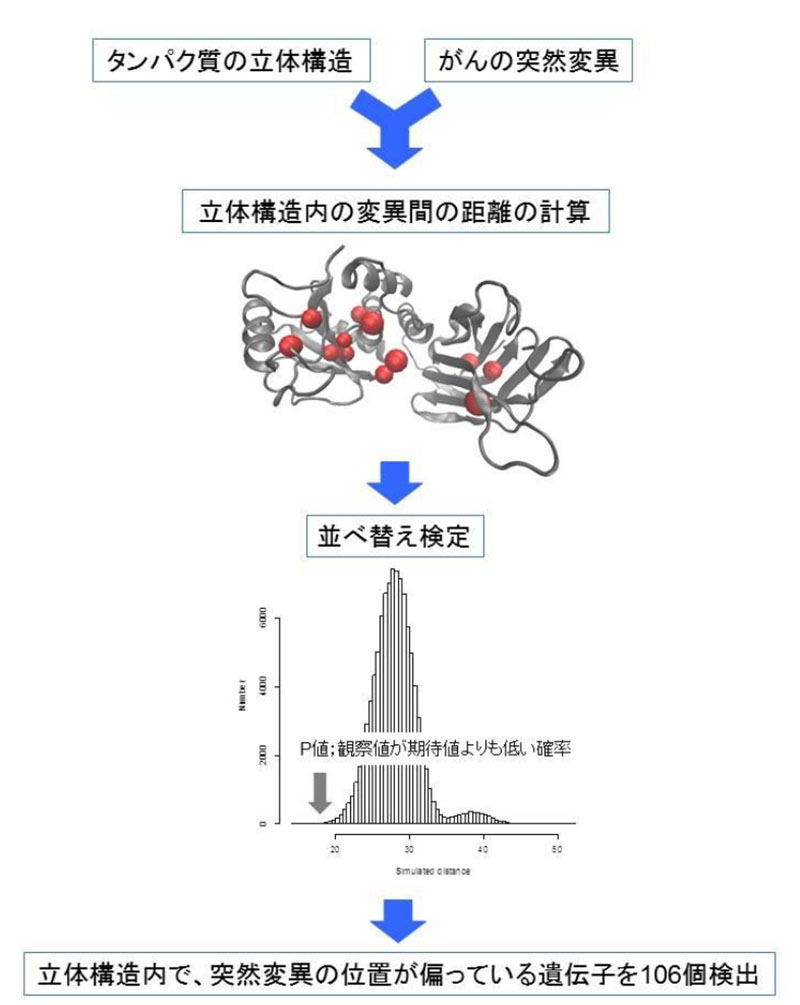
図:今回新たに開発したドライバー遺伝子検出手法
研究支援者やプロジェクト等
本研究の解析には、東京大学医科学研究所 ヒトゲノム解析センターのスーパーコンピュータ SHIROKANEを使用しました。また、研究の一部は公益社団法人 武田科学振興財団の支援を受けています。
書誌情報
【DOI】
http://dx.doi.org/10.1038/srep26483
【KURENAIアクセスURL】
http://hdl.handle.net/2433/214349
Akihiro Fujimoto, Yukinori Okada, Keith A. Boroevich, Tatsuhiko Tsunoda, Hiroaki Taniguchi & Hidewaki Nakagawa. (2016). Systematic analysis of mutation distribution in three dimensional protein structures identifies cancer driver genes. Scientific Reports 6:26483.
- 京都新聞(6月25日 10面)に掲載されました。





