植田充美 農学研究科教授、黒田浩一 同准教授、里村淳 同博士課程学生らの研究グループは、ゲノムワイドで高効率な編集技術を構築するために、DNA一本鎖切断酵素であるCas9 Nickaseを用いたCRISPR/Nickaseシステムを確立しました。
本研究成果は、2017年5月18日午後6時に英国の学術雑誌「Scientific Reports」に掲載されました。
研究者からのコメント
本研究では、真核生物のモデルである酵母細胞を用いて、ゲノムワイドで高効率な編集技術を構築するためにCRISPR/Nickaseシステムを確立しました。旧来のゲノム編集法であるCRISPR/Cas9システムでは全てのゲノム領域を編集することができない、非相同末端結合(DNA二本鎖切断において、切断末端同士が連結される修復)により標的配列に望まない塩基の挿入、欠失が生じてしまうという大きな欠陥が目立ってきておりますが、私たちの開発したCRISPR/Nickaseシステムによってこれらの欠陥を超越することができました。今後さらに、動物細胞にも展開することにより、ゲノム上のほぼすべてのSNPs(一塩基多型。DNAの塩基配列の中で、一塩基だけが標準と異なる個人差の一つ)の修復編集が可能になるとともに、逆に、目的通りのSNPsの導入も可能になり、これからのゲノム科学の発展に貢献できると考えております。
概要
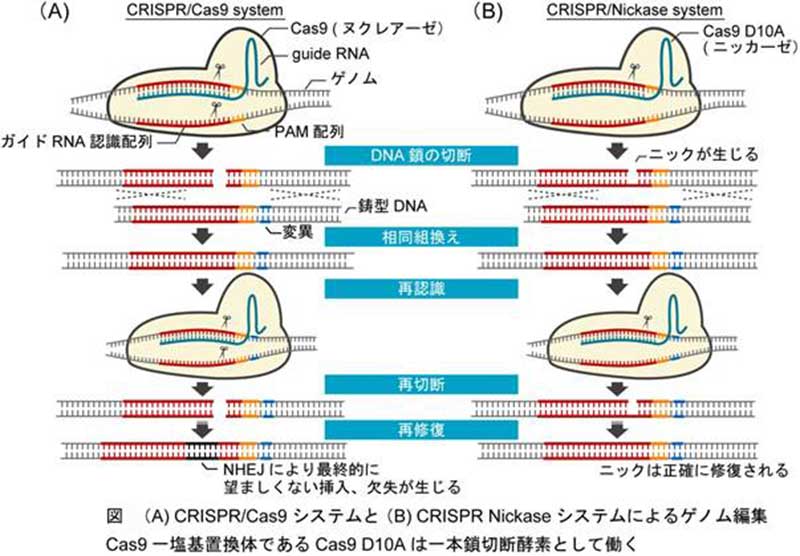
近年、ゲノム編集法としてCRISPR/Cas9システムが注目を集めています。このシステムでは、ガイドRNAがPAM配列(NGG:Nは任意の塩基)に続く20塩基を認識して、Cas9がDNAを切断します。切断領域に相補的なドナーDNAを加えると、相同組換え(DNAの塩基配列がよく似た部位(相同部位)で起こる組換え)により任意の配列をノックイン(挿入)できます。しかし、CRISPR/Cas9システムは全てのゲノム領域を編集することはできないという大きな欠点があります。また、ガイドRNA認識配列外の塩基を編集する場合、相同組換え後もガイドRNA認識配列が残るため、Cas9ヌクレアーゼにより、ゲノムが繰り返し切断・修復された結果、最終的には非相同末端結合により望まない塩基の挿入、欠失が生じてしまうという大きな欠陥もあります。
そこで本研究グループは、旧来法よりもはるかにゲノムワイドで高効率な編集技術を構築するために、一本鎖切断酵素であるCas9 Nickaseを用いました。Cas9 Nickaseで生じる一本鎖切断(ニック)は正確に修復されます。また旧来のCas9により生じる二本鎖切断と異なり、ニックは相同組換えを誘導しますが、非相同末端結合は誘導しません。そのため、Nickaseによる相同組換え後に標的配列が残った時でもニックが再度導入されるだけで、非相同末端結合による余分な配列の挿入や欠損は起こりません。また、本システムはニックから50塩基対離れた領域でも正確に編集できました。これは、本システムが編集可能塩基に制限があるCRISPR/Cas9よりも、理論上40%以上も広いゲノム領域、すなわち、ほぼゲノム全域を編集できることを示しています。
詳しい研究内容について
書誌情報
【DOI】 https://doi.org/10.1038/s41598-017-02013-7
【KURENAIアクセスURL】 http://hdl.handle.net/2433/224892
Atsushi Satomura, Ryosuke Nishioka, Hitoshi Mori, Kosuke Sato, Kouichi Kuroda & Mitsuyoshi Ueda (2017). Precise genome-wide base editing by the CRISPR Nickase system in yeast. Scientific Reports, 7, 2095.
- 朝日新聞(5月25日 23面)に掲載されました。





